Interactions hydrophobes
: entre groupes hydrophobiques comme les groupements cycliques de la phénylalanine et de la tyrosine. De telles interactions excluent les molécules d'eau.
Forces de Van der Waals
: il s'agit de dipôle temporaires de faible force.. Ceux-ci peuvent se former parce que les nuages électroniques des atomes individuels peuvent fluctuer, donnant naissance à des dipôles temporaires.
Ponts disulfures
: Nous l'avons vu dans le chapitre un: une cystéine oxydée peut former un lien covalent avec une autre cystéine. Ces ponts ne peuvent se produire spontanément que dans des conditions oxydantes (ce qui n'est pas le cas dans le cytoplasme, qui est légèrement réducteur). Les ponts disulfures se forment donc dans la lumière du réticulum endoplasmique chez les cellules eucaryotes, et dans l'espace périplasmique chez les bactéries. Le réticulum endoplasmique est également riche en une protéine facilitant la formation de ces ponts: il s'agit de la PDI, ou protein disulfide isomerase. Celle-ci peut oxyder des cystéines réduites et les forcer à former un pont disulfure. La PDI, sous sa forme réduite, peut également briser un pont disulfure déjà établi en formant un pont temporaire avec une des deux cystéines impliquées, ce qui permet à la deuxième cystéine d'établir un pont ailleurs. Ce processus permet de briser les ponts disulfures établis par accident entre deux cystéines qui n'auraient pas dû être pontées, et permet ainsi à la protéine de retrouver une structure tertiaire plus stable.
| Type d'interaction | Changement dans l'énergie libre (en kJ/mol) |
| lien covalent (carbone-carbone) | -350 (très solide!) |
| interaction hydrophobique | -10 |
| interaction ionique (pont de sel) | -4 |
| pont hydrogène (entre H et F,O ou N) | -3 |
| forces de Van der Waals | -1 |
2.3.2 Fibres et globules
On sépare souvent les protéines globulaires des protéines fibreuses.
2.3.2.1 Protéines globulaires
Les protéines globulaires ont une forme compacte, où on peut distinguer une surface en contact avec le solvant et un coeur qui en est isolé. Les résidus hydrophiles se retrouvent souvent à la surface; les résidus hydrophobes le plus souvent à l'intérieur.
Les ponts disulfures peuvent se former plus facilement à l'intérieur des protéines globulaires, parce qu'ils nécessitent des conditions oxydantes et que le cytoplasme est généralement réducteur à cause de la présence de glutathione. À l'abri des molécules d'eau qui les hydrateraient, les acides aminés chargés enfouis dans les protéines globulaires peuvent former des ponts de sel entre eux. Les acides aminés hydrophobes peuvent aussi plus facilement s'y empiler pour former des régions dépourvues d'eau.
2.3.2.2 Protéines fibreuses
Puisque toutes les protéines sont à la base une longue chaîne polypeptidique, on peut se les représenter comme des spaghettis. Alors qu'une protéine globulaire est une boule de spaghettis refroidis et figée, une protéine fibreuse s'étire en longueur.
Voici quelques protéines fibreuses typiques:
(a) la tropocollagène, la composante de base du collagène, qui est la protéine la plus abondante du corps humain. La structure secondaire la plus présente dans le tropocollagène est l'hélice de collagène, de type PPII et qui est gauchère. Cette hélice n'est pas stabilisée par des ponts hydrogènes internes.
(b) la kératine et la tropomyosine, retrouvées dans la peau et les muscles, respectivement. Les deux consistent d'hélices alpha, qui sont droitières, et sont stabilisées par des ponts hydrogènes internes orientés dans le même sens que la fibre, ce qui leur permet d'être étirées. Les ponts hydrogènes des hélices peuvent se briser sous la traction sans qu'il y ait de bris de liens covalents; quand la tension se relâche les ponts hydrogènes se reforment. Suffisamment étirée, la kératine peut aussi adopter une structure en feuillet beta.
Une protofibrille de kératine est formée par l'enroulement de deux ou trois hélices alpha les unes autour des autres, en tournant de façon gauchère. 11 protofibrilles forment une microfibrille de kératine; un grand nombre de telles microfibrilles forment une fibre de kératine.
(c) la fribroïne de la soie est composée de régions très rigides et riches en feuillets b antiparallèles. Ces régions sont séparées par des régions moins structurées conférant une certaine élasticité à la soie.
On peut retrouver dans la fibroïne une répétition du genre Gly-Ala-Gly-Ala-Gly-Ser avec la chaîne latérale de Gly un atome d'hydrogène) occupant entièrement l'une surface du feuillet:
Les régions rigides riches en feuillets b de la fibroïne sont peu élastiques parce que leurs nombreux ponts hydrogènes sont perpendiculaires à la direction de la chaîne polypeptidique. L’élasticité de la soie est conférée par les domaines amorphes séparant les feuillets beta.
2.3.3 Domaines
Il existe des structures tertiaires qui ont connu un tel succès que la nature les a utilisés comme domaines modulaires. Ces domaines peuvent se retrouver sur des protéines différentes pour y jouer le même rôle. Plusieurs types de domaines d'attache à l'ADN sont communs à de grandes familles de facteurs de transcription qui pourtant n'ont pas du tout les mêmes fonctions: les doigts de zinc, les domaines bZIP, les homéodomaines, les hélice-boucle-hélice, etc).
La plupart des domaines ont une structure stable qu'on peut transférer dans un autre contexte protéique. On peut ainsi fabriquer des protéines chimériques consistant de domaines de différentes origines. Un facteur de transcription très utilisé en laboratoire pour l'étude de l'activation de la transcription est une fusion du domaine d'attache à l'ADN de la protéine de levure Gal4 et du domaine d'activation de la transcription de la protéine virale VP-16.
2.3.3.1 bZIP, ou leucine zipper

Cette structure consiste en une hélice amphipathique contenant des leucines répétées régulièrement. Deux hélices de ce type peuvent interagir entre elles de façon à permettre la dimérisation de protéines, particulièrement celle de facteurs de transcription comme les membres de la famille AP-1 ou (comme dans la figure ci-contre) GCN4. Un domaine basique à la base des hélices permet l'interaction avec l'ADN (chargé négativement).
2.3.3.2 bHLH (hélice-boucle-hélice)

Ce domaine d'attache à l'ADN présente deux hélices reliées par une boucle non structurée. La structure permet la dimérisation de facteurs de transcription et l'attachement à l'ADN sur une séquence de type E box, CANNTG.
Les facteurs de transcription Myc et MyoD contiennent des domaines bHLH.
Dans cette figure, on nous présente un dimère de domaines bHLH liés à l'ADN.
2.3.3.3 Helix turn helix (ou hélice tourne hélice)
Voici le domaine hélice-tour-hélice du répresseur lambda.
Il s'agit de deux hélices alpha antiparallèles reliées entre elle par une simple boucle non structurée. Plusieurs protéines (et même plusieurs domaines) contiennent une telle structure. Le répresseur lac ou l'homéodomaine, par exemple, contiennent des domaines hélice-tourne-hélice.
2.3.3.4 Winged helix
Structure hélicale permettant la liaison à l'ADN pour certains facteurs de transcription, comme par exemple le facteur HNF-3.
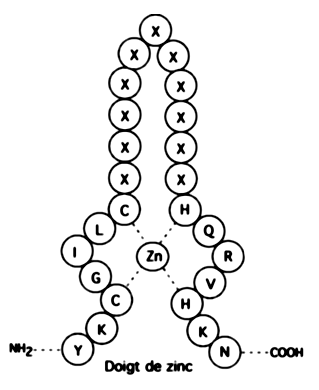
2.3.3.5 Zinc finger
|
Domaine d'attache à l'ADN caractérisé par une boucle à la base de laquelle se trouve des histidines ou des cystéines qui coordonnent un atome de zinc. La présence de zinc stabilise la structure. Les doigts de zinc sont des domaines d'interaction avec l'ADN ou entre protéines. On retrouve des doigts de zinc dans de très nombreux facteurs de transcription (SP1, Récepteurs nucléaires...) |
2.3.3.6 Homeobox
Ce domaine consiste en une séquence très conservée (chez les levures, les plantes aussi bien que chez les animaux) de 60 acides aminés formant trois hélices (voyez la partie homeobox du domaine POU, ci-dessous). Ce motif se retrouve dans un grand nombre de facteurs de transcription impliqués dans le développement embryonnaire. Il s'agit d'un domaine d'interaction avec l'ADN.
2.3.3.7 Domaine POU

|
POU est un acronyme de Pit-1, Oct-2 et Unc-86, trois facteurs de transcription chez qui le domaine a d'abord été identifié. Le domaine POU est lui-même constitué de deux sous-domaines: un domaine spécifique à la famille POU (le domaine POU-S), et un homéodomaine (ou homeobox). L'homéodomaine se retrouve chez un grand nombre de facteurs de transcription impliqués dans le développement. (Il a reçu son nom à cause de sa présence dans des facteurs de transcription dont l’inactivation entraînait des mutations homéotiques, du genre remplacement une antenne par une patte).
|
2.3.3.8 Domaine NFkB Le domaine d'attache à l'ADN assez complexe de la famille NFkB/rel évoque la forme d'un papillon.
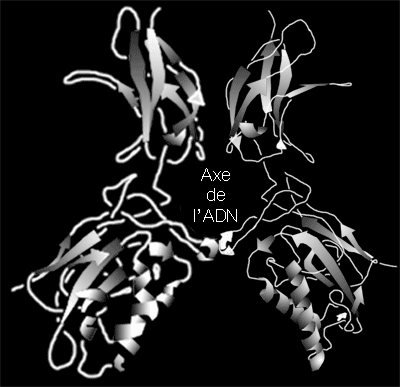
Contrairement à des domaines de petites taille comme le doigts de zinc, ces domaines-ci requièrent un grand échafaudage protéique pour se maintenir.
On retrouve ce motif dans des facteurs de transcription tels p50, p65, rel, et NFAT.
2.3.3.9 La boîte MADS ou MADS box
La boîte MADS est un motif commun à certains facteurs de transcription. Son nom vient des quatre premiers facteurs dans lesquels on l'a identifiée: MCM1, Agamous, Deficiens et SRF. On compte au dessus de cent facteurs contenant des boîte MADS, particulièrement parmi les facteurs de transcription qui dirigent la morphogénèse des plantes.
Domaine d'attache à l'ADN qui reconnaît la séquence YAAC(G/T)G. On retrouve des domaines SANT dans les facteurs de transcription v-myb, TFIIB" ainsi que d'autres protéines nucléaires comme Ada2, Swi3 et N-CoR.
2.3.3.10 domaine SANT
2.3.3.11 PHD finger
Motif s'apparentant au doigt de zinc, le doigt PHD se retrouve dans des protéines comme p300, CBP, TIF1, ash-1 (une protéine du groupe trithorax), l'histone acétyltransférase Moz, etc.
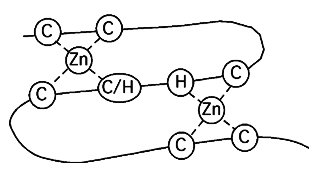
2.3.3.12 RING finger
|
"Real Interesting New Gene": voilà l'origine du nom "ring finger". Il s'agit d'une forme particulière de doigt de zinc.
On retrouve un tel Ring finger, par exemple, dans la protéine BRCA1 (dont la mutation est associée au cancer du sein), dans la sous-unité MAT1 du facteur de transcritpion TFIIH, ou dans plusieurs membres des ubiquitine-transférases de la famille E3. |
|
Une variété de RING finger est le motif RBCC, qui contient un RING finger, deux domaines supplémentaires de liaison de Zinc (les B-box), une hélice de leucine (Coiled Coil) et une région C-terminale variable). Ce domaine permet lui aussi une interaction protéine-protéine.
2.3.3.13 Bromodomaine
Le bromodomaine compte 110 acides aminés et se retrouve dans plusieurs protéines associées à la chromatine, des levures aux animaux; on le retrouve dans les acétyltransférases Gcn5, P/CAF, CBP, p300, TAFII250, etc. Certaines protéines contiennent des bromodomaines très modifiées, avec des parties manquantes ou remplacées par d'autres séquences. On croit le bromodomaine impliqué dans certaines interactions entre protéines et il pourrait jouer un rôle dans l'assemblage des complexes menant à l'activation de la transcription.
2.3.3.14 Chromodomaine
Identifié à cause d'une région d'homologie entre les protéines d'hétérochromatine HP1 et Polycomb, ce domaine a été ainsi nommé en raison de sa fonction présumée (qui serait de jouer un rôle dans la restructuration de la chromatine). Le chromodomaine consiste en une cinquantaine de résidus organisés sous forme de trois feuillets beta pressés contre une hélice alpha. Le chromodomaine de la protéine HP1 lui permet de se lier à une histone H3 méthylée sur la lysine 9. (Cette méthylation est effectuée par une autre protéine à chromodomaine, SUV39H1). On sait que dans certains cas, le chromodomaine peut interagir avec de l'ARN; on soupçonne d'ailleurs qu'une telle interaction ARN-chromodomaine joue un rôle dans l'établissement de régions hétérochromatiques par le système du RNAi (ou "RNA interference").
2.3.3.15 Domaine riche en glutamine, domaine riche en proline, domaine acidique
Vous rencontrerez probablement ces termes si vous étudiez les domaines d'activation des facteurs de transcription. Des facteurs comme SP1 ont en effet un domaine d'activation riche en glutamine (domaine qui, d,ailleurs, ne fonctionne pas chez un eucaryote primitif comme S. cerevisiae); des facteurs comme GAL4, VP16 ou GCN4 ont des domaines riches en acides aminés acides; des facteurs comme CTF1 ont des domaines d'activation riches en proline. On doit donc se souvenir de ces particularités quand on analyse une séquence en acides aminés et qu'on y cherche une région fonctionnelle.
2.3.3.16 Répétitions WD40

|
Les répétitions WD40 sont caractérisées par un motif Tryptophane-acide aspartique qui se répète à tous les 40 résidus environ. Ce dom aine semble permettre l'interaction entre certaines protéines. On le retrouve dans de nombreux types de protéines, incluant des facteurs de transcription (Taf72 chez S. pombe), des répresseurs (Hir1p de S. cerevisiae) ou même des cyclines (Cdc20 chez l'humain). C'est un motif assez courant. |
Pour en savoir plus sur différents domaines comme le domaine 14-3-3 de nombreuses kinases, je vous recommande le site suivant.
2.3.3.17 T-box

La boîte T ou T-box a été caractérisée en raison de sa présence dans le facteur de transcription Brachyury (T). On la retrouve dans plusieurs facteurs de transcription impliqués dans le développement, les décisions précoces quant à l'identité cellulaire, l'établissement du plan corporel et l'organogénèse. La T-box est un domaine assez grand (17-26kDa), très riche en feuillets beta, et qui représente souvent une proportion considérable de la protéine totale. Les facteurs possédant une T-box lient la séquence TCACACCT. La structure de la T-box peut être visualisée sur ce site.
2.3.4 Les chaperones
Les protéines adoptent spontanément leur structure tertiaire. Certaines sont si stables que même après une dénaturation complète elles retrouvent spontanément et rapidement leur structure normale (c'est le cas de la RNAse A, une nucléase qu'on peut faire bouillir à volonté et qui retrouve toujours son activité par la suite).
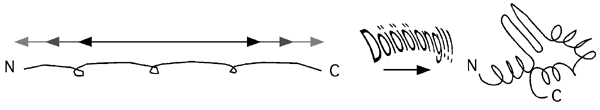
Toutes les protéines ne sont pas dans ce cas, bien sûr: pour la plupart, un traitement dénaturant aura résulté en leur précipitation pure et simple ou du moins en une agrégation à cause d'interactions hydrophobiques entre résidus exposés par le dépliement de la protéine.
Il existe cependant une classe de molécules qui aident les protéines à retrouver leur structure tertiaire si elles viennent à la perdre. Ces molécules sont les chaperones, qui protègent les protéines d'un repliement inadéquat ou de l'agrégation. Les protéines HSP (heat shock proteins) par exemple, servent à protéger les protéines des dégâts causés par un choc thermique. Elles peuvent aussi aider les protéines à retrouver leur forme momentanément perdue: c'est par exemple le cas de Hsp60, qui aide au transport de protéines dans la matrice mitochondriale. L'enzyme F1 ATPase est synthétisé sous forme d'un précurseur à partir d'un gène nucléaire. Ce précurseur contient un peptide signal lui permettant d'être reconnu par un récepteur membranaire des mitochondries afin de faciliter son importation. Cependant, la F1 ATPase sous sa forme repliée est trop grosse pour passer par le canal de transport mitochondrial. Elle doit d'abord être dépliée par l'action d'enzymes idoines. L'ATPase, maintenant dépliée, peut ensuite passer par le canal et se retrouver dans la matrice mitochondriale. Là, une protéase coupe son peptide signal et la Hsp60 l'aide à retrouver sa forme initiale. Sans Hsp60, l'ATPase ne réussit pas à reprendre sa structure tertiaire et à rejoindre le complexe où elle agit d'habitude.
Chez E. coli, on connait bien le système GroES-GroEL qui sert à replier correctement les structures diformes. 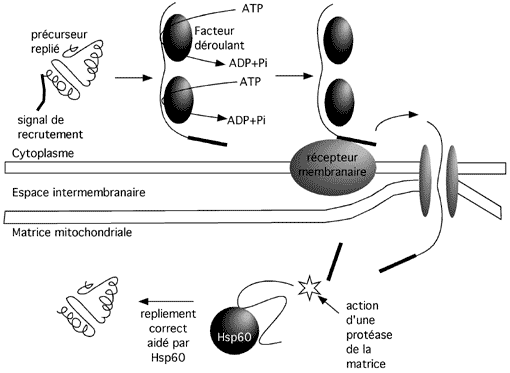
GroES est un anneau de 10 KDa consistant en sept sous-unités; GroEL est aussi un anneau de sept protéines mais pesant 58 KDa.
En l'absence de GroES, deux anneaux GroEL s'empilent l'un sur l'autre. Le substrat mal plié entre dans la cavité centrale d'un des anneaux. GroES vient alors couvrir le trou, et induit dans la cavité un profond changement de conformation: elle passe de majoritairement hydrophobe à majoritairement hydrophile, ce qui facilite aussi un changement de conformation dans le substrat. Ce changement donne la chance au substrat de peut-être retrouver une conformation stable et appropriée. Après vingt secondes, quoiqu'il advienne, le substrat est relâché -qu'il soit bien replié ou non; il peut toujours, si ça n'a pas marché, recommencer le cycle.
Voici un fait intéressant ayant rapport aux chaperones: dans le cas de la fibrose kystique, l'origine de la maladie est une (ou plusieurs) mutations dans le gène CFTR, qui code pour une pompe à ions Cl-. En bousculant la circulation des ions de part et d'autre de la membrane plasmique des cellules pulmonaires, la maladie entraîne un problème d'hydratation dans les poumons et facilite l'accumulation de mucus dans lequel poussent des bactéries opportunistes.
Ces mutations dans le gène CFTR sont accompagnées par le remplacement de certains acides aminés par d'autres dans la protéine pendant sa traduction. Dans la majeure partie des cas, les mutations ponctuelles n'interfèreraient pas avec le fonctionnement de la pompe à ions, mais malheureusement les chaperones reconnaissent la protéine comme étant mal formée et ne lui permettent pas de se rendre jusqu'à la membrane. Les patients sont donc victimes de leur propre système de contrôle de la qualité protéique.
2.3.5 Quand la structure tertiaire cafouille
La maladie de la vache folle, une encéphalite spongiforme transmissible à l'homme, nous a sensibilisés à l'impact que peut avoir sur la santé la structure tertiaire d'une protéine.
|
L'hypothèse du prion, qui valut le prix Nobel à son inventeur, le Dr Stanley Prusiner, suggère que l'agent pathologique de cette maladie (comme celle d'autres encéphalites spongiformes, dont le nom vient du fait qu'elles changent le cerveau en véritable éponge pleine de trous, et qui incluent la maladie de Creutzfeld-Jakob, le syndrome de Gerstmann-Strausler-Scheinker et l'insomnie familiale fatale) n'est pas une bactérie, ni un virus, ni même quoique ce soit contenant un acide nucléique. Ce serait une protéine à laquelle on a donné le nom de prion.
|
 Dr Stanley Prusiner Photo (c) 2000 The Nobel Institute. |
La structure primaire du prion est codée par le gène PRNP (pour "PRIon Protein"). Chez un individu sain, la protéine codée par ce gène n'a rien de dangereux et fait partie de notre bagage habituel de protéines. On désigne cette protéine normale par le nom de PrPC; il s'agit d'une protéine extra-cellulaire fixée à l'extérieur de la membrane plasmique par une ancre de GLI (ou glycosyl-phosphatidylinositol.
Chez une victime de la maladie, la séquence primaire ne change pas: c'est la structure tertiaire qui est profondément modifiée, passant d'une structure principalement faites d'hélices alpha en une structure très riche en feuillets beta. Cette nouvelle structure est extrêmement stable et résiste à presque toutes les techniques de désinfection. La protéine ayant adoptée cette nouvelle structure est appelée PrPSC, pour "scrapie", le nom anglais de la tremblante du mouton, le pendant ovin de la maladie de la vache folle.
Comment la structure passe-t-elle de PrPC à celle de PrPSC? C'est là que l'histoire devient vraiment bizarre: il semble que la conversion soit catalysée par PrPSC elle-même. À la manière d'un vampire, PrPSC entre en contact avec un PrPC sain et le convertit en PrPSC.
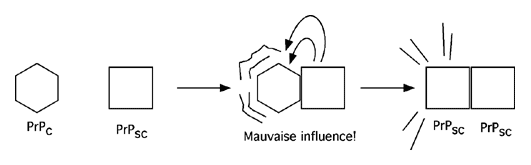
Le PrPC converti en PrPSC est indifférentiable du premier PrPSC et va à son tour convertir d'autre PrPC. On assiste alors à une réaction en chaîne de conversion vers PrPSC. PrPSC s'accumule dans les cellules, et il semble qu'il finisse par former de gros agrégats. Il détruit en tout cas les cellules du cerveau, entraînant les sévères troubles neurologiques que l'on connaît et éventuellement la mort. Il existe aussi une forme d'origine génétique de la maladie. En effet, certaines mutations dans le gène PRNP favorisent l'adoption de la conformation PrPSC plutôt que la conformation habituelle PrPC. Chez les individus porteurs de ces mutations, la traduction de l'ARNm de PRNP peut donner naissance à une particule PrPSC. Et dès que celle-ci est synthétisée par le ribosome, elle peut se mettre à convertir PrPC en plus de PrPSC.

(Une autre forme d'exemple de propagation des prions,
qui a bercé notre enfance! (c) Éditions Dupuis).
Ce phénomène n'est pas limité aux mammifères: chez la levure, le produit du gène URE2 semble lui aussi avoir une forme PrPC comme une forme PrPSC. Les prions semblent donc être une forme extrêmement avancée de parasites moléculaires: non contents de puis er dans nos cellules le matériel requis à leur reproduction, comme le font les virus, ils ne se donnent même pas la peine d'emporter leur matériel génétique avec eux.
2.3.6 Épissage protéique
Il existe chez les protéines un phénomène analogue à celui de l'épissage chez les ARN messagers: une partie de la chaîne polypeptidique peut être enlevée quelque part entre ses deux extrémités, et les deux bouts restant être raccordées ensemble.
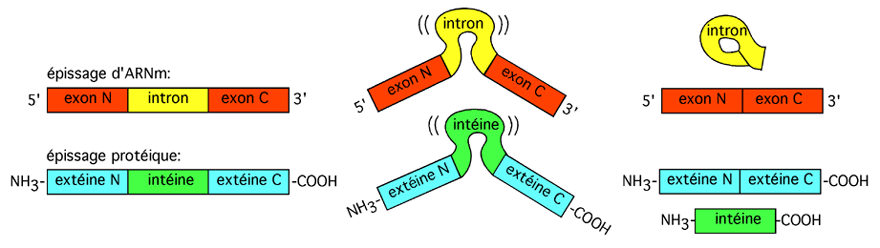
Les intéines et les extéines sont à ces protéines épissées ce que les introns et les exons sont à l'ARNm. (Mais ne nous mêlons pas: les exons ne codent pas pour des extéines, ni les introns pour des intéines! Il ne s'agit que d'un parallèle de principe).
Ce sont les intéines elles-mêmes qui se chargent de leur excision à partir du précurseur avec lequel elles ont été synthétisées. On en a recensé au-dessus de 150 à ce jour, chez différents procaryotes et chez la levure.
| Voici le mécanisme d'excision d'une intéine. On peut souligner une parenté avec le domaine autocatalytique de la protéine de signalisation Hedgehog, qui se coupe en deux pour greffer à sa partie N-terminale une molécule de cholestérol lui permettant de se fixer à la membrane cellulaire. |
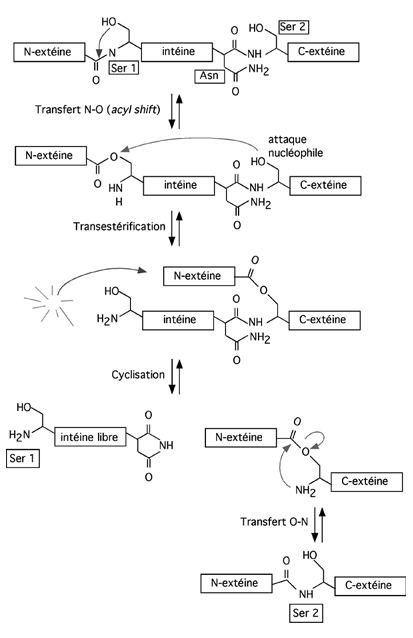
|
Les Dr Chilkoti et Filipe (le dernier de l'université McMaster en Ontario) ont mis au point une technique de purification des protéines utilisant une intéine. Dans leur approche, la séquence d'une intéine est introduite entre celle de l'ELP (Elastin-like polypeptide) et celle de la protéine d'intérêt. La protéine de fusion finale, à cause de la présence de l'ELP, précipite en présence de sel ou à une température inférieure à 30-40°C, menant à un très grand enrichissement (puisque les autres protéines, elles, restent solubles). La protéine de fusion récupérée et resuspendue est ensuite incubée de façon à ce que l'intéine se coupe elle-même, libérant la protéine d'intérêt.
À c e sujet, voir les références suivantes: Banki et al. (2005) Nat Methods 2: 659-62 et Ge et al. (2005) J Am Che Soc 127: 11228-9.
Une catégorie d'intéines a une particularité intéressante: une fois excisées du précurseur, ces protéines constituent des endonucléases qui reconnaissent des sites de restriction particulièrement longs. On les appelle des méganucléases.
Il existe une certaine similarité entre ces méganucléases et l'endonucléase HO responsable de la recombinaison somatique entre les locus HML, HMR et MAT chez la levure.
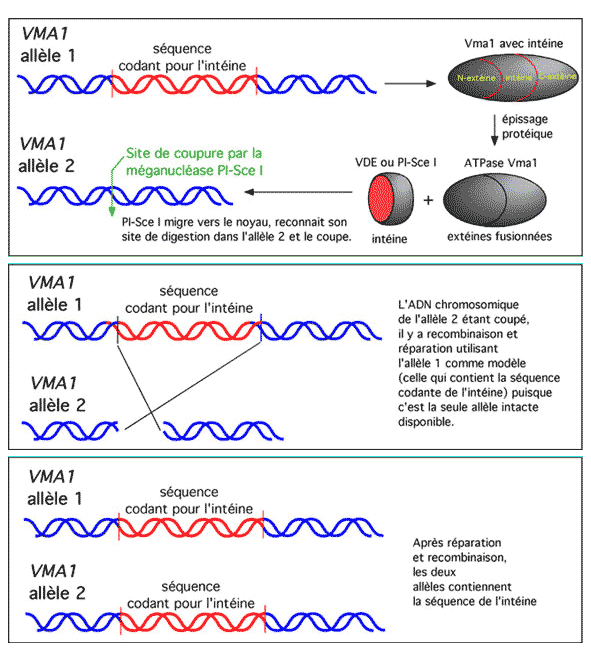
Un aspect encore plus intéressant de ces endonucléases est qu'elles jouent un rôle dans la propagation de leur propre séquence codante. Voici comment, en utilisant comme exemple la méganucléase PI-SceI, qui est une intéine retrouvée au beau milieu du polypeptide d'une ATPase de levure, Vma1.
Il existe deux types d'allèle pour VMA1. L'une d'entre elles contient la séquence codante pour PI-SceI. L'autre allèle ne contient pas cette séquence codante, mais à la place contient le site de restriction reconnu par la méganucléase PI-Sce I:
5' ATCTATGTCGGGTGC GGAGAAAGAGGTAATGAAATGGCA 3'
3' TAGATACAGCC CACGCCTCTTTCTCCATTACTTTACCGT 5'
|
Quand la levure, dans sa forme diploïde, contient les deux allèles, l'une d'entre elles conduit à la production de PI-Sce I. La nucléase une fois libérée de son précurseur, Vma1, migre vers le noyau où elle va retrouver son site de restriction qui justement se trouve dans l'allèle de VMA1 ne contenant pas l'insertion codant pour PI-Sce I. (Ce voyage vers une cible précise explique le nom anglais de ce type de nucléases: homing endonucleases).
La nucléase va couper ce site. Avec un chromosome coupé en deux, la levure va essayer de réparer les dégâts en utilisant l'autre allèle par recombinaison homologue. Cette réparation va dupliquer l'insertion codant pour PI-Sce I dans le site ouvert par la nucléase, et après la réparation son gène aura été introduit dans l'allèle qui auparavant n'en disposait pas. PI-Sce I agit donc comme une sorte de parasite moléculaire, qui veille à la propagation de sa propre séquence codante dans les génomes qui en sont dépourvus. |
|
2.3.6.2 Épissage protéique par le protéasome
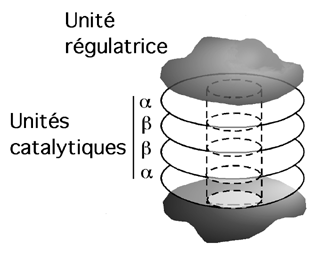
Nous verrons dans le chapitre 3 que le protéasome consiste en une usine moléculaire de dégradation des protéines. Cette usine est elle-même formée de plusieurs sous-unités protéiques formant un tunnel par lequel passent les protéines destinées à la dégradation.
|
Structure générale d'un protéasome. Les unités régulatrices contrôlent l'accès à un tunnel formé par les sous-unités catalytiques a et b. Chacune de ces sous-unités sont elles-mêmes formées de sept protéines formant un anneau. L'empilage des quatre anneaux forment un tunnel dans lequel ont lieu les réactions enzymatiques catalysées par le protéasome. |
|
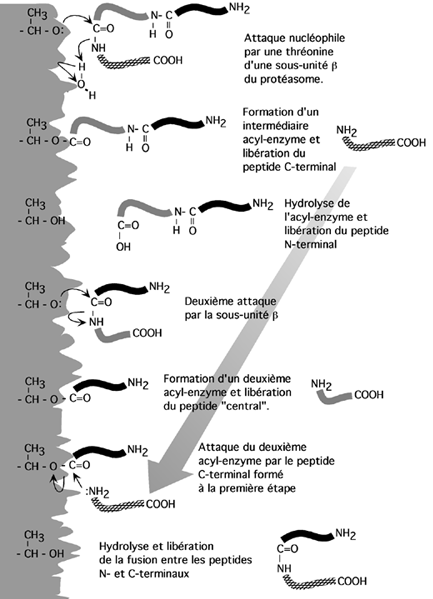
On a récemment découvert que le protéasome était non seulement responsable de la dégradation, mais qu'il pouvait aussi être impliqué dans une forme d'épissage protéique. Les lymphocytes T de type CD8+ reconnaissent des antigènes que génèrent les cellules présentant l'antigène (APC). Ces antigènes consistent en peptides de 8 à 10 résidus, et proviennent des produits de dégradation des protéines- cibles par le protéasome. Ces résidus se retrouvemt donc normalement à la queue-leu-leu dans la protéine originale. On a cependant découvert il y a peu que ces peptides peuvent consister en deux courtes séquences de résidus normalement séparées par une séquence intercalaire, et fusionnées par le protéasome. Le mécanisme de cette fusion est décrit ci-contre et correspond bien à une forme d'épissage protéique. |
|
| Retour à la page d'accueil | Section précédente | Section suivante |