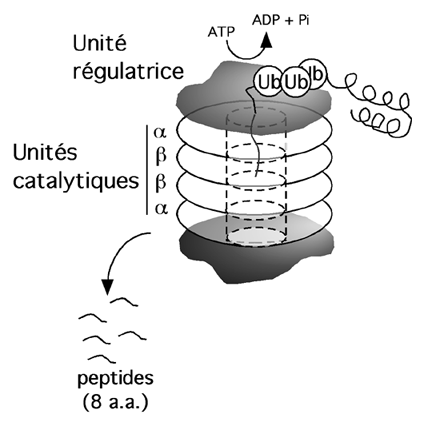
Au protéasome 26S, la protéine est déroulée par des ATPases et est dégradée en peptides de 8 acides aminés environ.
| Retour à la page d'accueil | Section précédente | Section suivante |
3.5 Ajout de polypeptides
3.5.1 ubiquitination
Voir les revues suivantes sur l'ubiquitination: Pickart CM. Mechanisms underlying ubiquitination. Annu Rev Biochem. 2001;70:503-33 ainsi que Voges D, Zwickl P, Baumeister W. The 26S proteasome: a molecular machine designed for controlled proteolysis. Annu Rev Biochem. 1999;68:1015-68 et Hershko A, Ciechanover A. The ubiquitin system. Annu Rev Biochem. 1998;67:425-79.
L'ubiquitine est une petite protéine de 76 acides aminés, extrêmement conservée dans le monde vivant. Son rôle principal consiste à marquer une protéine pour la dégradation par le protéasome 26S. Son importance a valu à ses découvreurs le prix Nobel de chimie en 2004.

|
L'ubiquitine est ajoutée par sa propre extrémité C-terminale à des résidus lysine des protéines-cibles ou, parfois, à l'extrémité N-terminale de certains facteurs (MyoD ou LMP-1 de EBV, p.e.). D'autres molécules d'ubiquitine peuvent ensuite être ajoutées par leur C-terminal à la lysine K48 de la précédente pour former une longue chaîne qui permet l'accostage de la protéine sur des sites de reconnaissance localisés sur la particule régulatrice du protéasome 26S.
Au protéasome 26S, la protéine est déroulée par des ATPases et est dégradée en peptides de 8 acides aminés environ. |
Trois familles d'enzymes sont requises pour ubiquitiner les protéines (E1 à E3); un nouvel enzyme, E4, semble faciliter l'allongement de la chaîne poly-ubiquitine. Au moins quatre ubiquitines sont nécessaires pour signaliser la destruction de la protéine. Les membres des familles E2 et E3 forment des complexes qui s'associent avec la protéine-cible; E3 fournit l'association à la cible et E2 fournit l'ubiquitine.
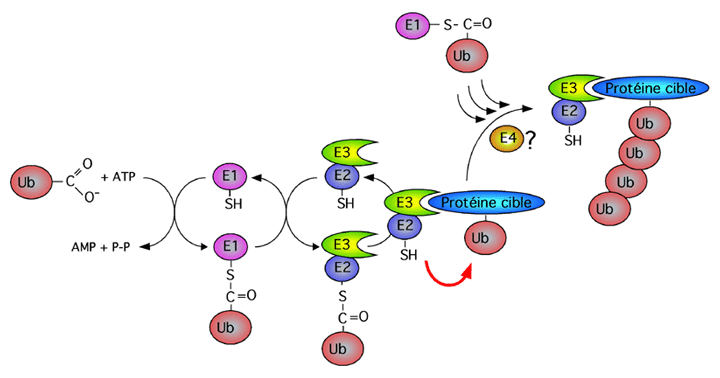
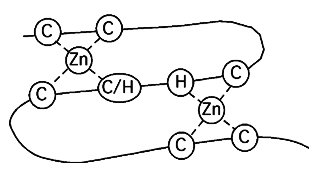
Le signal d'ubiquitination des protéines cibles n'est pas très bien défini, quoiqu'on ait décrit une "destruction box" qui en fait office chez les cyclines. Sa séquence est très dégénérée et ressemble à ceci: R (A/T) (A) L (G) X (I/V) (G/T) (N), où les résidus entre parenthèses sont présents 50% du temps ou plus. En outre, certaines modifications post-traductionnelles (comme la phosphorylation) peuvent changer une séquence peptidique inactive en signal de reconnaissance pour le système de dégradation; c'est ce qui se passe dans le système IkB-NFkB illustré plus bas.
3.5.2 La règle du N-terminal (ou "N-end rule") et le dégron.
La "règle du N-terminus" (ou "N-end rule"), spécifie que certains résidus, quand ils occupent la place N-terminale d'un peptide, peuvent donner un signal de destruction. Les acides aminés concernés, qu'on peut identifier dans la table ci-dessous, se combinent avec une lysine située légèrement en aval pour former une cassette appelée N-degron. Le N-degron est reconnu par un complexe protéique E2-E3 qui ajoute des ubiquitines à la lysine. La protéine est ensuite détruite par le protéasome comme nous venons de le voir.
On a utilisé ce dernier phénomène pour développer un système d'auto-destruction qui permet d'étudier le fonctionnement de certaines protéines en forçant leur dégradation à des moments donnés. Ce système repose sur le clonage, en phase avec le cadre de lecture de la protéine d'intérêt, d'une cassette (appelée dégron, un petit nom très technologique) qui peut recruter le système d'ubiquitination en réponse à un choc thermique.
Demi-vie d'une fusion Ubiquitine-Résidu-β-Galactosidase (en minutes)
Résidu
E.coli
S. cerevisiae
Arginine, Arg, R 2 2
Lysine, Lys, K 2 3
Phenylalanine, Phe, F 2 2
Leucine, Leu, L 2 2
Tryptophane, Trp, W 2 2
Tyrosine, Tyr, Y 2 10
Histidine, His, H Plus de 600 3
Isoleucine, Ile, I Plus de 600 30
Acide aspartique, Asp, D Plus de 600 3
Acide glutamique, Glu, E Plus de 600 30
Asparagine, Asn, N Plus de 600 3
Glutamine, Gln, Q Plus de 600 10
Cysteine, Cys, C Plus de 600 Plus de 1200
Alanine, Ala, A Plus de 600 Plus de 1200
Serine, Ser, S Plus de 600 Plus de 1200
Threonine, Thr, T Plus de 600 Plus de 1200
Glycine, Gly, G Plus de 600 Plus de 1200
Valine, Val, V Plus de 600 Plus de 1200
Proline, Pro, P On ne sait pas, puisque la proline ne participe pas au dégron.
Methionine, Met, M Plus de 600 Plus de 1200
(D'après Varshavsky, PNAS 93: 12142-12149, 1996)
Le dégron de levure représenté ci-bas contient
(a) la séquence de l'ubiquitine,
(b) une arginine, et
(c) la séquence d'une dihydrofolate réductase (DHFR) thermosensible.
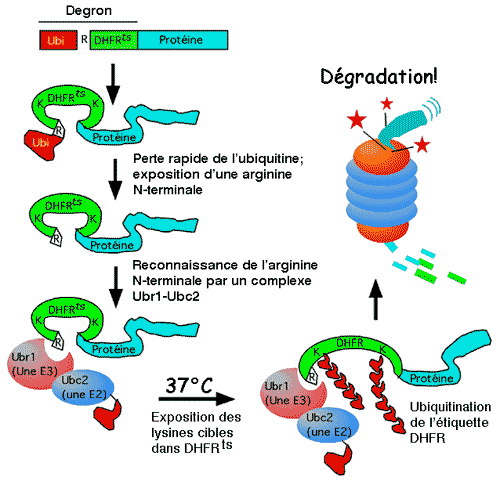
L'ubiquitine située en N-terminus est enlevée pendant ou tout juste après la traduction, un phénomène normal et attribuable à des Ubiquitines-protéases. Ces protéases coupent juste après la séquence de l'ubiquitine, laissant exposé l' acide aminé suivant (sauf s'il s'agit d'une proline). Dans le cas qui nous occupe ici, la coupure de l'ubiquitine expose une arginine N-terminale. Cette arginine est reconnue par un complexe E2-E3 (Ubc2-Ubr1, ce dernier produit s'appelant aussi N-recognin) qui ne peut pas ubiquitiner les lysines de la DHFR voisine tant que la température reste basse. Lors d'un choc thermique, la DHFR subit un changement conformationnel et les lysines-cibles sont exposées, menant à leur ubiquitination puis à la dégradation de la protéine entière.
L'ubiquitine ne sert pas qu'à marquer les protéines pour la dégradation. De courtes chaînes d'ubiquitine jouent probablement un rôle différent (Les histones H2A, H2B et H1 peuvent être ubiquitinées dans leur partie C-terminale sans être condamnées à la destruction). Par exemple, on sait que la protéine de réparation de l'ADN Rad26 aide à ubiquitiner la lysine 123 de l'histone H2B. Cette ubiquitination semble être un pré-requis à la méthylation de la lysine 4 de l'histone H3, et on soupçonne que cela est dû au recrutement de la sous-unité 19S du protéasome (la sous-unité régulatrice) par H2B-K123-Ubi. La méthylation de H3K4 permet le recrutement de SAGA via sa sous-unité Chd1, et SAGA peut ensuite procéder à l'acétylation de l'histone H3 via sa sous-unité Gcn5.
Le facteur de transcription NFkB est contrôlé par un système d'ubiquitination et de phosphorylation. NFkB est normalement maintenu dans le cytoplasme par son association avec un répresseur appelé IkB (inhibiteur de kappa B) riches en répétitions ankyrines ou avec des formes immatures de NFkB contenant ces répétitions. Suite à un signal pro-inflammatoire approprié , IkB est phosphorylé; cette modification le rend apte à être ubiquitiné. Le protéasome dégrade alors le répresseur et NFkB est libre de migrer vers le noyau et son site d'attache sur l'ADN.
.gif)
3.5.2 Sumoylation
Il existe des protéines apparentées à l'ubiquitine, soit par leur séquence, soit par leur capacité d'être greffée à des protéines déjà existentes. C'est le cas des protéines SUMO ( small ubiquitin-like modifier).La sumoylation est une variante de l'ubiquitination. Elle implique l'attachement de SUMO à une lysine présente dans divereses cibles comme p53, IκBα, ou PML. Elle ne mène pas à l'assemblage de longues chaînes de SUMO et ne mène pas non plus à la dégradation. La sumoylation est réversible.
SUMO semble augmenter la stabilité de ses cibles et moduler leur compartimentalisation cellulaire. Pour les facteurs inhibiteurs IκB et Mdm2, SUMO semble faire compétition à l'ubiquitine et pourrait agir comme stabilisateur.
AOS1 et UBA2 agissent de concert comme enzyme E1 pour la sumoylation. Ces deux protéines présentent une homologie avec les parties N-terminale et C-terminale de UBA1, l'enzyme E1 impliquée dans l'ubiquitination.
Retour à la page d'accueil Section précédente
Section suivante