BCM-514
Biochimie des protéines
Les acides aminés
1.5 Le code génétique
Les acides aminés sont assemblés en chaîne selon un ordre déterminé par une séquence d'ADN. Chaque acide aminé est représenté sur l'ADN par un triplet de bases appelé codon.
Puisqu'il existe quatre nucléotides dans l'ADN, il existe aussi une possibilité maximale de 43 codons, ou 64. Il n'y a cependant que 20 acides aminés, qui se partagent ces possibilités entre eux (et avec trois signaux d'arrêt de la traduction). La distribution des codons n'est pas égale: il n'y a par exemple qu'un seul codon codant pour le tryptophane mais six pour l'arginine.
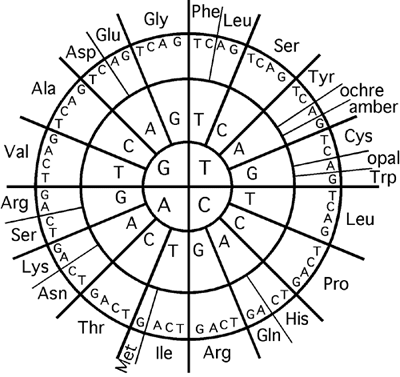
|
Le code génétique. Chaque codon (ou triplet de nucléotides) se lit du centre vers l'extérieur. En plus des codons désignant les 20 acides aminés, il existe trois codons STOP, qui marquent la fin de la séquence d'une protéine: les codons amber (TAG), ochre (TAA) et opal (TGA)
|
1.5.1 Universalité relative du code génétique
Le code génétique est extrêmement conservé dans la nature. Des plus humbles procaryotes aux plus massifs séquoias, les acides aminés et les codons stop sont presque toujours les mêmes. Il existe cependant quelques mutations qui se sont accumulées au cours de l'évolution, principalement chez certaines mitochondries qui ont leur propre génome, et dont le code peut varier un peu face à celui utilisé par l'ADN nucléaire.
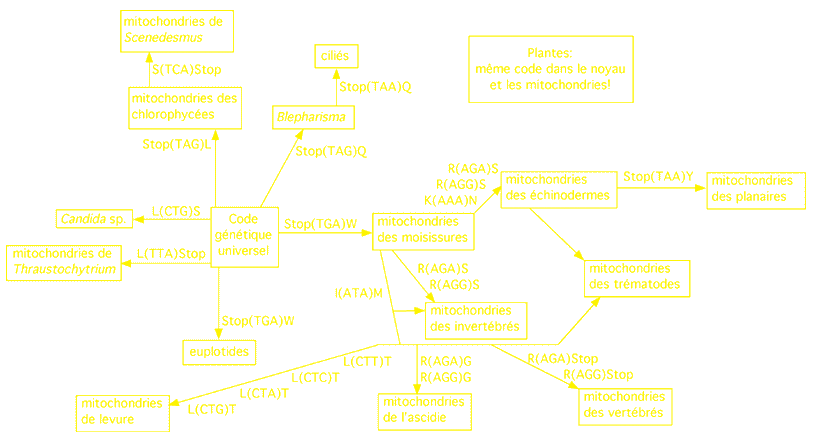
Variations sur un thème: certains codons ont changé de signification dans certaines organelles ou certains organismes au cours du temps. Les flèches indiquent des mutations cumulatives qui peuvent refléter soit une conservation évolutive, soit une évolution parallèle. Ainsi, les mitochondries des planaires cumulent les mutations Stop(TGA)W, R(AGA)S, R(AGG)S, K(AAA)N et Stop (TAA)Y. Notez que chez les plantes, noyaux et mitochondries utilisent le même code et peuvent donc s'échanger des gènes.
(Adapté des travaux de Weberndorfer, Hofacker et Stadler)
1.5.2 Utilisation préférentielle des codons
Il est utile en biotechnologie de se rappeller que différents organismes n'utilisent pas avec le même enthousiasme tous les codons disponibles pour un même acide aminé. Certains codons sont clairement favorisés par rapport à d'autres, ce qui se reflète souvent dans l'abondance des ARNt correspondants. Ce phénomène est probablement lié à des considérations économiques pour les organismes.
En exprimant une séquence d'ADN dans un système hétérologue (H. sapiens dans E. coli, par exemple), mieux vaut donc être sûr d'utiliser une séquence qui sera facilement traduite par les ARNt de l'hôte, quitte à changer par génie génétique des codons défavorisés par des codons favorisés. Sinon, on risque de s'exposer à des tracas du genre
- traduction retardée entraînant une baisse de la stabilité de l'ARNm;
- terminaison prématurée, pouvant mener à des protéines trop courtes ou instables;
- changements de cadre de lecture ou mauvaise incorporation;
- inhibition de la synthèse protéique.
Dans la liste ci-dessous, on compare trois organismes et leur préférence en fait de codons codant pour l'arginine. Si tout était égal par ailleurs, on s'attendrait à ce que chacun des six codons Arg soit représenté avec une fréquence d'environ 16,5% et en effet, chez l'être humain, il n'existe qu'un léger biais qui défavorise le codon CGT. Par contre, chez S.cerevisiae, les codons CGG, CGC et CGA ne représentent respectivement que 2%, 4% et 5% des codons Arg du génome et la levure ne fabrique que très peu des ARNt portant les anticodons appropriés. Une séquence humaine exprimée dans la levure aurait intérêt à voir tous ses codons Arg changés pour AGA, qui est fortement favorisé par cet organisme.
|
codon
|
H. sapiens
|
S. cerevisiae
|
E. coli
|
|
AGG
|
21,5
|
17,0
|
3,2
|
|
AGA
|
21,0
|
54,0
|
4,3
|
|
CGG
|
18,2
|
2,0
|
9,0
|
|
CGA
|
10,7
|
5,0
|
5,0
|
|
CGT
|
9,1
|
17,0
|
36,0
|
|
CGC
|
19,5
|
4,0
|
40,0
|
1.5.2.1 Utilisation des codons: comparaison entre différentes espèces
(source: http://www.kazusa.or.jp/codon/
Homo sapiens (codon, acide aminé, fréquence sur 1).
|
UUU
|
F
|
0.46
|
|
UCU
|
S
|
0.18
|
|
UAU
|
Y
|
0.44
|
|
UGU
|
C
|
0.45
|
|
UUC
|
F
|
0.54
|
|
UCC
|
S
|
0.22
|
|
UAC
|
Y
|
0.56
|
|
UGC
|
C
|
0.55
|
|
UUA
|
L
|
0.07
|
|
UCA
|
S
|
0.15
|
|
UAA
|
stop
|
0.28
|
|
UGA
|
stop
|
0.5
|
|
UUG
|
L
|
0.13
|
|
UCG
|
S
|
0.06
|
|
UAG
|
stop
|
0.22
|
|
UGG
|
W
|
1.0
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
CUU
|
L
|
0.13
|
|
CCU
|
P
|
0.28
|
|
CAU
|
H
|
0.41
|
|
CGU
|
R
|
0.08
|
|
CUC
|
L
|
0.20
|
|
CCC
|
P
|
0.33
|
|
CAC
|
H
|
0.59
|
|
CGC
|
R
|
0.19
|
|
CUA
|
L
|
0.07
|
|
CCA
|
P
|
0.27
|
|
CAA
|
Q
|
0.26
|
|
CGA
|
R
|
0.11
|
|
CUG
|
L
|
0.40
|
|
CCG
|
P
|
0.11
|
|
CAG
|
Q
|
0.74
|
|
CGG
|
R
|
0.21
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
AUU
|
I
|
0.36
|
|
ACU
|
T
|
0.24
|
|
AAU
|
N
|
0.46
|
|
AGU
|
S
|
0.15
|
|
AUC
|
I
|
0.48
|
|
ACC
|
T
|
0.36
|
|
AAC
|
N
|
0.54
|
|
AGC
|
S
|
0.24
|
|
AUA
|
I
|
0.16
|
|
ACA
|
T
|
0.28
|
|
AAA
|
K
|
0.42
|
|
AGA
|
R
|
0.21
|
|
AUG
|
M
|
1.0
|
|
ACG
|
T
|
0.12
|
|
AAG
|
K
|
0.58
|
|
AGG
|
R
|
0.20
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
GUU
|
V
|
0.18
|
|
GCU
|
A
|
0.26
|
|
GAU
|
D
|
0.46
|
|
GGU
|
G
|
0.16
|
|
GUC
|
V
|
0.24
|
|
GCC
|
A
|
0.40
|
|
GAC
|
D
|
0.54
|
|
GGC
|
G
|
0.34
|
|
GUA
|
V
|
0.11
|
|
GCA
|
A
|
0.23
|
|
GAA
|
E
|
0.42
|
|
GGA
|
G
|
0.25
|
|
GUG
|
V
|
0.47
|
|
GCG
|
A
|
0.11
|
|
GAG
|
E
|
0.58
|
|
GGG
|
G
|
0.25
|
Saccharomyces cerevisiae Triplet, acide aminé, fréquence (sur 1)
|
UUU
|
F
|
0.59
|
|
UCU
|
S
|
0.26
|
|
UAU
|
Y
|
0.56
|
|
UGU
|
C
|
0.63
|
|
UUC
|
F
|
0.41
|
|
UCC
|
S
|
0.16
|
|
UAC
|
Y
|
0.44
|
|
UGC
|
C
|
0.37
|
|
UUA
|
L
|
0.28
|
|
UCA
|
S
|
0.21
|
|
UAA
|
stop
|
0.47
|
|
UGA
|
stop
|
0.3
|
|
UUG
|
L
|
0.29
|
|
UCG
|
S
|
0.10
|
|
UAG
|
stop
|
0.23
|
|
UGG
|
W
|
1.0
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
CUU
|
L
|
0.13
|
|
CCU
|
P
|
0.31
|
|
CAU
|
H
|
0.64
|
|
CGU
|
R
|
0.15
|
|
CUC
|
L
|
0.06
|
|
CCC
|
P
|
0.15
|
|
CAC
|
H
|
0.36
|
|
CGC
|
R
|
0.06
|
|
CUA
|
L
|
0.14
|
|
CCA
|
P
|
0.42
|
|
CAA
|
Q
|
0.69
|
|
CGA
|
R
|
0.07
|
|
CUG
|
L
|
0.11
|
|
CCG
|
P
|
0.12
|
|
CAG
|
Q
|
0.31
|
|
CGG
|
R
|
0.04
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
AUU
|
I
|
0.46
|
|
ACU
|
T
|
0.35
|
|
AAU
|
N
|
0.59
|
|
AGU
|
S
|
0.16
|
|
AUC
|
I
|
0.26
|
|
ACC
|
T
|
0.21
|
|
AAC
|
N
|
0.41
|
|
AGC
|
S
|
0.11
|
|
AUA
|
I
|
0.27
|
|
ACA
|
T
|
0.30
|
|
AAA
|
K
|
0.58
|
|
AGA
|
R
|
0.48
|
|
AUG
|
M
|
1.0
|
|
ACG
|
T
|
0.14
|
|
AAG
|
K
|
0.42
|
|
AGG
|
R
|
0.21
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
GUU
|
V
|
0.39
|
|
GCU
|
A
|
0.38
|
|
GAU
|
D
|
0.65
|
|
GGU
|
G
|
0.47
|
|
GUC
|
V
|
0.21
|
|
GCC
|
A
|
0.22
|
|
GAC
|
D
|
0.35
|
|
GGC
|
G
|
0.19
|
|
GUA
|
V
|
0.21
|
|
GCA
|
A
|
0.29
|
|
GAA
|
E
|
0.71
|
|
GGA
|
G
|
0.22
|
|
GUG
|
V
|
0.19
|
|
GCG
|
A
|
0.11
|
|
GAG
|
E
|
0.29
|
|
GGG
|
G
|
0.12
|
Escherichia coliTriplet, acide aminé, fréquence (sur 1)
|
UUU
|
F
|
0.57
|
|
UCU
|
S
|
0.15
|
|
UAU
|
Y
|
0.57
|
|
UGU
|
C
|
0.44
|
|
UUC
|
F
|
0.43
|
|
UCC
|
S
|
0.15
|
|
UAC
|
Y
|
0.43
|
|
UGC
|
C
|
0.56
|
|
UUA
|
L
|
0.13
|
|
UCA
|
S
|
0.12
|
|
UAA
|
stop
|
0.63
|
|
UGA
|
stop
|
0.29
|
|
UUG
|
L
|
0.13
|
|
UCG
|
S
|
0.15
|
|
UAG
|
stop
|
0.08
|
|
UGG
|
W
|
1.0
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
CUU
|
L
|
0.1
|
|
CCU
|
P
|
0.16
|
|
CAU
|
H
|
0.57
|
|
CGU
|
R
|
0.38
|
|
CUC
|
L
|
0.1
|
|
CCC
|
P
|
0.12
|
|
CAC
|
H
|
0.43
|
|
CGC
|
R
|
0.40
|
|
CUA
|
L
|
0.04
|
|
CCA
|
P
|
0.19
|
|
CAA
|
Q
|
0.35
|
|
CGA
|
R
|
0.06
|
|
CUG
|
L
|
0.5
|
|
CCG
|
P
|
0.53
|
|
CAG
|
Q
|
0.65
|
|
CGG
|
R
|
0.10
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
AUU
|
I
|
0.51
|
|
ACU
|
T
|
0.17
|
|
AAU
|
N
|
0.45
|
|
AGU
|
S
|
0.15
|
|
AUC
|
I
|
0.42
|
|
ACC
|
T
|
0.43
|
|
AAC
|
N
|
0.55
|
|
AGC
|
S
|
0.28
|
|
AUA
|
I
|
0.07
|
|
ACA
|
T
|
0.13
|
|
AAA
|
K
|
0.76
|
|
AGA
|
R
|
0.04
|
|
AUG
|
M
|
1.0
|
|
ACG
|
T
|
0.27
|
|
AAG
|
K
|
0.24
|
|
AGG
|
R
|
0.02
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
GUU
|
V
|
0.26
|
|
GCU
|
A
|
0.16
|
|
GAU
|
D
|
0.63
|
|
GGU
|
G
|
0.34
|
|
GUC
|
V
|
0.22
|
|
GCC
|
A
|
0.27
|
|
GAC
|
D
|
0.37
|
|
GGC
|
G
|
0.40
|
|
GUA
|
V
|
0.15
|
|
GCA
|
A
|
0.21
|
|
GAA
|
E
|
0.69
|
|
GGA
|
G
|
0.11
|
|
GUG
|
V
|
0.37
|
|
GCG
|
A
|
0.36
|
|
GAG
|
E
|
0.31
|
|
GGG
|
G
|
0.15
|
Les six codons les moins utilisés chez E.coli, et leur fréquence chez d'autres organismes (nombre par millier de codons):
|
|
AGG
|
AGA
|
CGA
|
CUA
|
AUA
|
CCC
|
|
|
R
|
R
|
R
|
L
|
I
|
P
|
|
E. coli
|
1,4
|
2,1
|
3,1
|
3,2
|
4,1
|
4,3
|
|
H. sapiens
|
11
|
11,3
|
6,1
|
6,5
|
6,9
|
20,3
|
|
D. melanogaster
|
4,7
|
5,7
|
7,6
|
7,2
|
6,9
|
18,6
|
|
C. elegans
|
3,8
|
15,6
|
11,5
|
7,9
|
9,8
|
4,3
|
|
S. cerevisiae
|
9,3
|
21,3
|
3
|
13,4
|
17,8
|
6,8
|
|
P. falciparum
|
4,1
|
20,2
|
0,5
|
15,2
|
33,2
|
1
|
|
C. pasteurianus
|
2,4
|
32,8
|
0,8
|
6
|
52,5
|
8,5
|
|
P. honkoshii
|
30,3
|
20,4
|
1
|
18
|
44,9
|
10,1
|
|
T. aquaticus
|
13,7
|
1,4
|
1,4
|
3,2
|
2
|
43
|
|
A. thaliana
|
10,9
|
18,4
|
6
|
9,8
|
12,6
|
5,2
|
(Strategies Newsletter (2000) vol. 3 (1) p.31)
Les huit codons les moins utilisés:
|
E.coli
|
Levure
|
Drosophile
|
Humain
|
acide aminé
|
|
AGG
|
AGG
|
|
|
R
|
|
AGA
|
|
AGA
|
|
R
|
|
AUA
|
|
AUA
|
|
I
|
|
CUA
|
|
|
CUA
|
L
|
|
CGA
|
CGA
|
CGA
|
CGA
|
R
|
|
CGG
|
CGG
|
CGG
|
|
R
|
|
CCC
|
|
|
CCG
|
P
|
|
UCG
|
|
|
|
S
|
|
|
CGC
|
|
CGU
|
R
|
|
|
CCG
|
|
|
P
|
|
|
CUC
|
|
CUU
|
L
|
|
|
GCG
|
|
GCG
|
A
|
|
|
ACG
|
|
ACG
|
T
|
|
|
|
UUA
|
UUA
|
L
|
|
|
|
GGG
|
|
G
|
|
|
|
AGU
|
|
S
|
|
|
|
UGU
|
|
C
|
|
|
|
|
GUA
|
V
|
|
|
|
|
UUG
|
L
|
Certaines souches bactériennes commerciales contiennent des transgènes exprimant les ARNt normalement sous-exprimés. De telles souches nous permettent de contourner le problème sans avoir à optimiser les codons des protéines que l'on veut faire exprimer.
BL21 (DE3) CodonPlus-RIL R (AGG, AGA), I(AUA) et L (CUA)
BL21 (DE3) CodonPlus-RP R (AGG, AGA) et P (CCC)
Rosetta ou Rosetta (DE3) R (AGG/AGA), R (CGG), I (AUA), L (CUA), P (CCC) et G (GGA)
(Les bactéries CodonPlus sont des produits de la compagnie Stratagene; Rosetta est un produit de la compagnie Novagen).
1.5.3 Le 21e et le 22e acide aminé
|
Après des décennies de travail avec nos vingt acides aminés familiers, il fut surprenant de découvrir qu'il en existait finalement d'autres qui ne relevaient pas d'une modification post-traductionnelle des résidus classiques mais qui étaient insérés dans les protéines en tant qu'acides aminés originaux à part entière, avec leur propre ARN de transfert. Le 21e acide aminé découvert (en 1986) est la selenocystéine (Sec), le 22e (en 2002) est la pyrrolysine. Ils ne sont quand même pas très courants.
Ces deux acides aminés sont codés par des codons STOP qui sont "contournés" par des systèmes spéciaux. L'ARNt de la selenocystéine reconnait le codon UGA; celui de la pyrrolysine le codon UAG. L'ARNt de la selenocysteine ne peut être synthétisés que si la cellule a accès à une source de selenium.
En outre, on sait que la séquence de l'ARN en aval de l'UGA (sur environ 40 bases) joue en rôle dans l'identité du UGA en tant que codon Sec plutôt que codon stop. Ce n'est donc pas n'importe quel codon UGA qui codera pour une selenocystéine. |
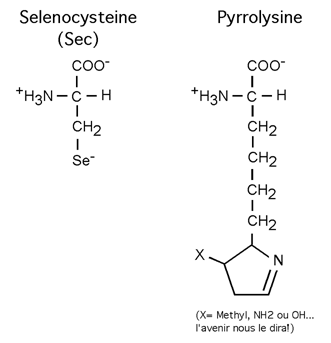
|
On a isolé la selenocystéine chez différents organismes eucaryotes et procaryotes; elle se retrouve dans des enzymes ayant besoin de selenium pour fonctioner, comme la glutathione peroxydase et la formate déhydrogénase chez les eucaryotes. La pyrrolysine, elle, a tout d'abord été découverte dans la monométhylamine méthyltransférase de l'archéa Methanosarcina barkeri.
La pyrrolysine est chargée directement sur son ARNt par une ARNt synthétase, mais dans le cas de la sélénocystéine la situation est un peu plus complexe.
Chez E. coli, quatre gènes sont requis pour produire une selenoprotéine: SelA, SelB, SelC et SelD.
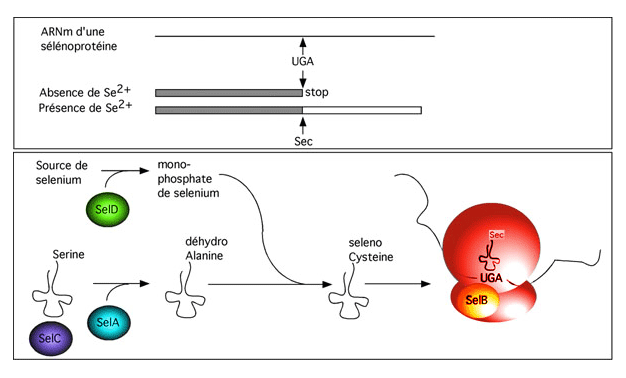
SelA code pour une selenocysteine synthase qui catalyse la conversion de seryl-tRNA en selenocysteyl-tRNA.
SelB code pour un facteur d'élongation analogue à EF-Tu mais avec une spécificité pour l'insertion de selenocysteine au codon UGA. Il nuit à la terminaison de la traduction au codon UGA, permettant à la selenocystéine de s'intégrer et à la traduction de se poursuivre le long de l'ORF étendu. SelD code pour une selenophosphate synthetase qui catalyse la réduction du selenium en selenium monophosphate. Ce phosphate est la source de métal pour la selenocystéine. SelC code pour l'ARNt de la selenocystéine, avec un anticodon UCA. Cet ARNt est d'abord chargé d'une sérine par la séryl-tRNA synthétase. C'est le plus long des ARNt connus jusqu'ici chez E. coli. Cette exception à la règle des vingt acides aminés souligne d'abord que rien n'est jamais coulé dans le béton en biologie, et ensuite que la nature a toujours une surprise ou deux en réserve pour nous quand nous croyons avoir tout compris.
1.5.4 Mutations au cours des siècles (et des millions d'années)!
Il est bien connu que des mutations s'accumulent dans le matériel génétique au cours des éons, et ces mutations génétiques dirigent la synthèse de protéines également mutées (et encore heureux, car sinon nous serions tous encore des petites morves qui nagent et rien de plus).
La mutation d'un codon dans une protéine passe par deux filtres: celui de l'occasion et celui de la sélection. Tout d'abord, plus une mutation est simple et plus elle a de chance de se produire (AGG muté en AGC est plus fréquent que AGG muté en TTT, parce qu'un seul nucléotide a été affecté). Ensuite, pour que la mutation soit maintenue, il faut qu'elle confère un avantage quelconque à son hôte, ou au moins qu'elle ne lui nuise pas.
La plupart de nos protéines sont là depuis très longtemps (le cytochrome C, par exemple, est un modèle qui dure et dure et dure). Comme ces protéines ancestrales remplissent très bien leur office, on n'y observe pas beaucoup de mutations d'une espèce à l'autre; les mutations ont tendance à être conservatrices (celles n'ont pas beaucoup d'impact). Si un résidu lysine joue un rôle important dans une protéine à cause de sa charge positive, sa mutation en arginine (même charge) sera plus facilement tolérée qu'une mutation en acide aspartique (charge inverse).
On peut utiliser le taux de mutations entre des protéines partagées par de nombreux représentants du monde vivant pour aider à déterminer leur lien de parenté. Dans la figure ci-dessous, on peut suivre les mutations les plus fréquemment observées dans des protéines aparentées. La direction des flèches indique celle des mutations; les flèches pointillées sont des mutations plus rares. Notez que pour ces mutations, il est toujours possible de passer d'un codon codant pour un premier acide aminé à un codon codant pour un deuxième en ne changeant qu'un seul nucléotide. Parallèlement à cette relation qui explique que ces mutations soient plus fréquentes parce que plus faciles à effectuer, on remarque que les chaînes latérales des résidus mutés ont une certaine parenté avec celles des résidus originaux (petit groupe pour petit groupe, comnme G vers A, ou encore charge négative pour charge négative comme D vers E et vice-versa, ou encore aromatique pour aromatique comme F vers Y et vice-versa); cela signifie probablement que la mutation n'a pas introduit d'élément néfaste au bon fonctionnement de la protéine, et n'a donc pas subi de pression évolutive tendant à la faire disparaître.
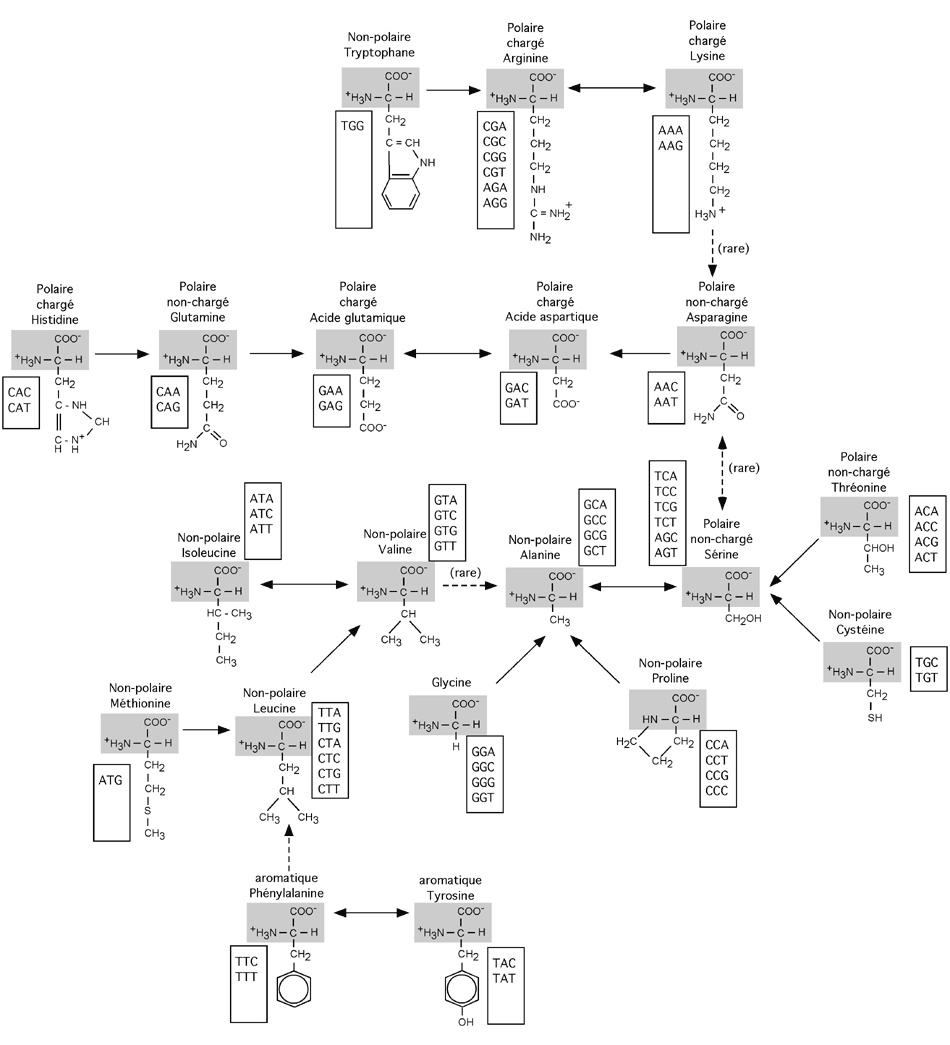
Les mutations les plus fréquemment observées.
Source: Current Protocols in Molecular Biology, Appendix , John Wiley & Sons, Toronto
benoit.leblanc@usherbrooke.ca




